Ny forskning fra QGG fokuserer på bedre dyrevelfærd for malkekvæg
Forskere ved Center for Kvantitativ Genetik og Genomforskning, Institut for Molekylærbiologi og Genetik ved Aarhus Universitet har lokaliseret DNA varianter, der kan give bedre yversundhed og dyrevelfærd for malkekvæg.
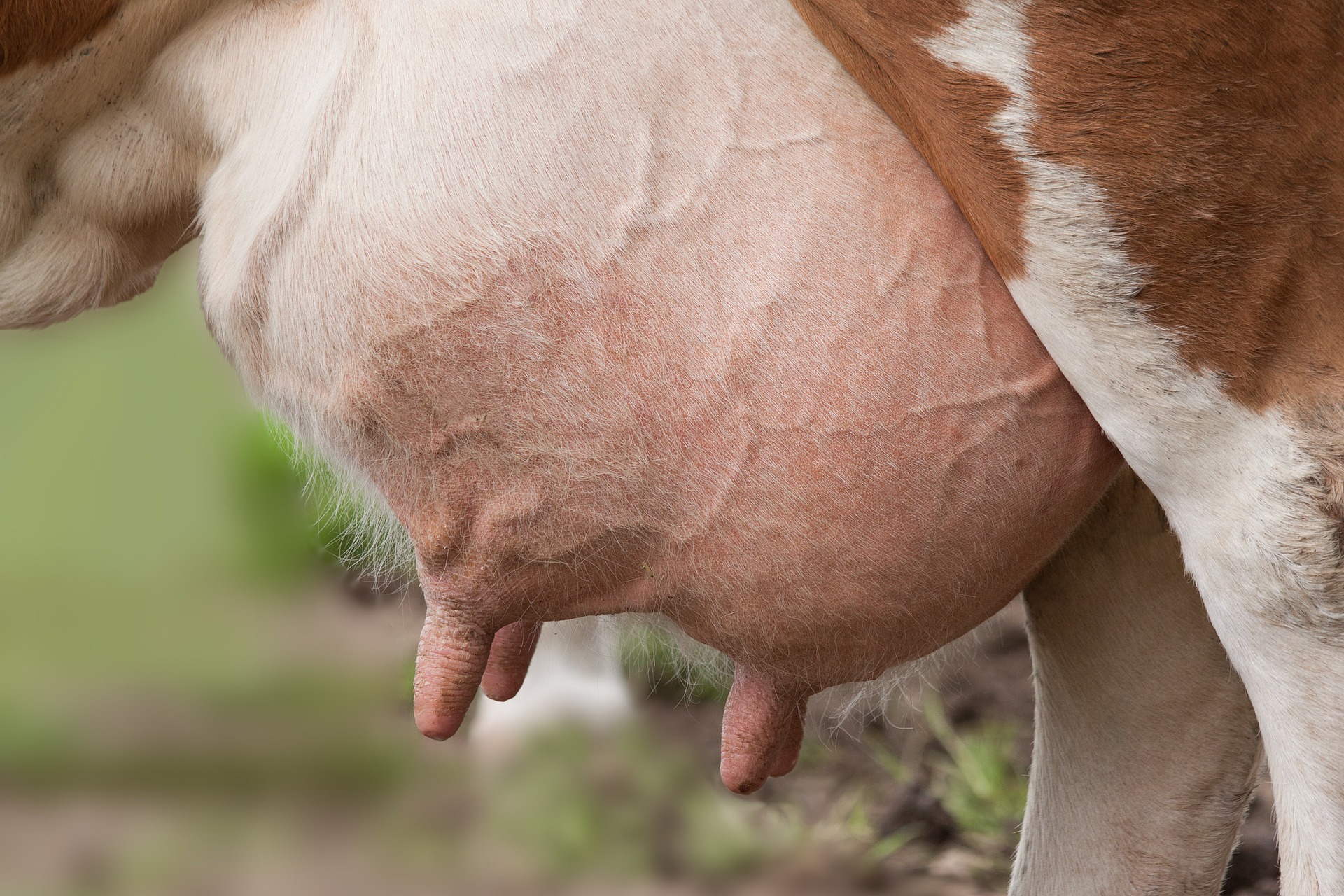
Det er i alles interesse at forbedre dyrevelfærden og reducere nødvendigheden af antibiotika i husdyrhold. Hos malkekvæg er yverbetændelse blandt de mest almindelige sygdomme og koster landmænd verden over store summer hvert år til dyrlægebesøg og medicin. Det er grunden til, at forskere i dyregenetik arbejder på at forbedre den generelle, genomiske selektion af malkekøer, med henblik på at avle individer med en større genetisk resistens mod yverbetændelse. Bedre yversundhed vil reducere landmandens udgifter, forøge malkekoens velfærd og give bedre mælk til forbrugerne.
I artiklen slår forskerne fast at en kos malkningsevne er defineret af malkningshastigheden, den gennemsnitlige mælkestrømningshastighed, den maksimale mælkestrømningshastighed og den totale malkningstid. Malkningsevnen har indflydelse på den arbejdstid der kræves til malkningen, og har en genetisk sammenhæng med forekomsten af yverbetændelse.Hvis en ko ikke tømmes ordentligt, når den bliver malket, er der højere risiko for yverbetændelse. Det er derfor en høj prioritet for forskerne og industrien at få kortlagt de gener, der fremmer genetisk resistens mod yverbetændelse.
I dette studie brugte forskerne genetisk information fra 4921 tyre af racen Nordisk Holstein, der tidligere er blevet genotypekortlagt med en BovineSNP50 bead chip tabel. Men denne chip dækker kun omkring 50.000 DNA variationer, jævnt spredt ud over det totale tyregenoms millioner af basepar. For at kompensere for dette bruger forskerne en teknik kaldet imputation. Ved hjælp af statistiske følgeslutninger udfylder imputationsteknikken de ’sorte huller’ mellem de allerede kendte genotyper, der er blevet kortlagt ved hjælp af SNP50 chippen. Dette giver et bredere, statistisk funderet billede af hele genomet. I dette studie blev de allerede kendte genotyper derfor imputeret først til high-density genotyper og efterfølgende til en helgenomsekvens.
Forskerne udførte efterfølgende flere analyser, hvor de specifikt ledte efter markører for egenskaberne yverindeks*) og malkningshastighed. Analyserne inkluderede en multiegenskabs-metaanalyse for de to egenskaber. De stærkeste signaler for yverindeks og malkningshastighed blev fundet på kromosomerne 19 og 20. Dette passer med tidligere fund og indikerer, hvor forskerne skal lede videre efter gener, der fremmer malkekøers resistens mod yverbetændelse.
De vigtige fund i dette studie kan udnyttes til en mere præcis prædiktion i den rutinemæssige, genomiske selektion. For avlere og landmænd med malkekvæg betyder dette bedre yversundhed og dermed bedre dyrevelfærd og lavere omkostninger til antibiotika og dyrlægeassistance.
*) I artiklen angiver forskerne, at ‘yverindekset beskriver det genetiske potentiale for yverets formning og er en lineær kombination af underindicier for yverets vedhæftning fortil, højden på yveret bagtil, bredden på yveret bagtil, yverkløft/støtte, yverdybde, patlængde, pattykkelse, placering af patter fortil og bagtil, samt yverbalance’.
Forskningen er udført ved det QGG-baserede Center for Genomisk Selektion i Dyr og Planter (GenSAP)
Mere information:
Link til den videnskabelige artikel i Journal of Dairy Science
