Nye resultater fra forbedrede metoder og modeller for SNP-baseret heritabilitet og selektionsanalyser
I en nyligt publiceret artikel beskriver professor Doug Speed fra Center for Kvantitativ Genetik og Genomforskning (QGG) sammen med Anubhav Kaphle og David Balding (University of Melbourne) deres nyeste resultater inden for komplekse træks genetiske arkitekturer, og hvordan deres arbejde med forbedrede metoder og modeller har korrigeret de oprindelige metoder, der kom frem for mere end ti år siden.
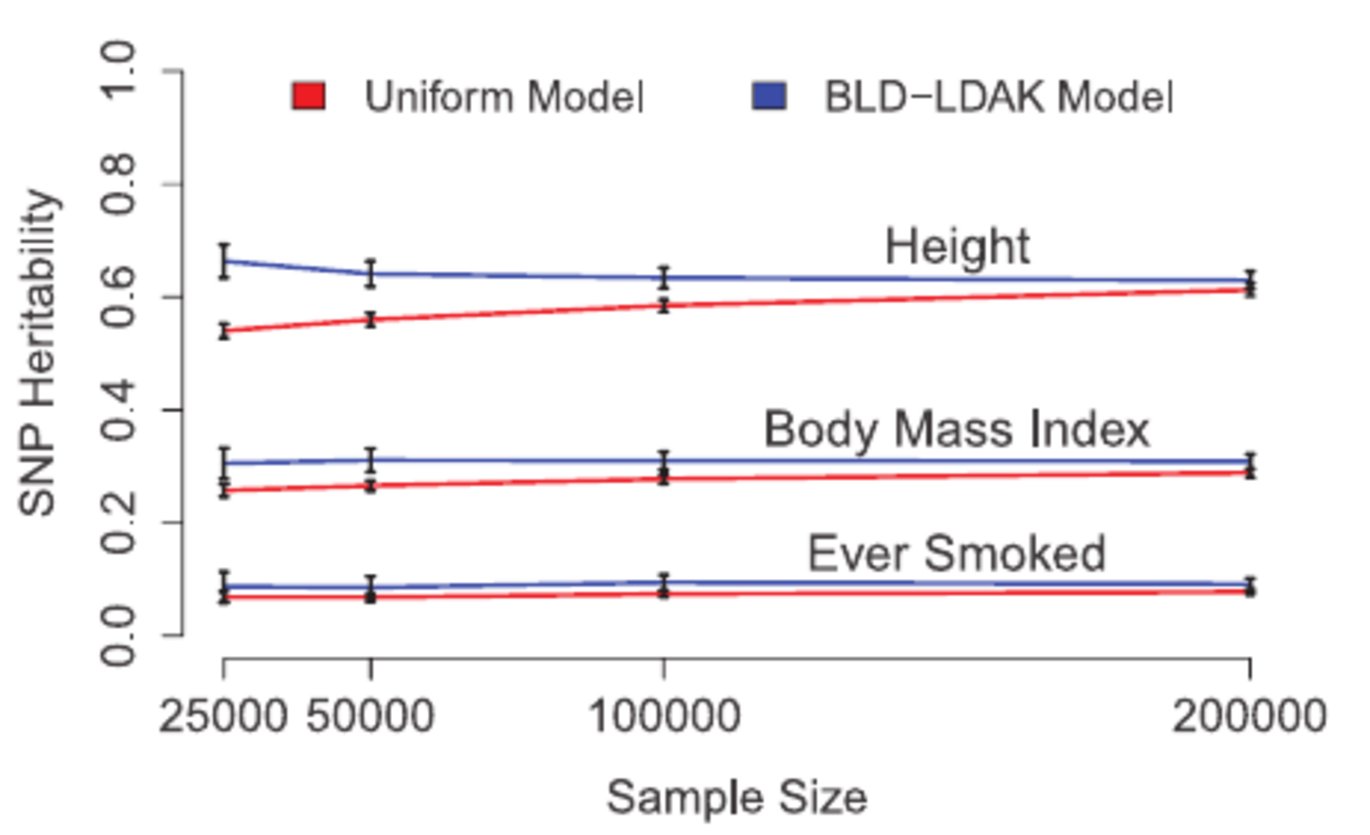
Artiklen præsenterer nye resultater fra analyser af data fra UK Biobank. Helt konkret viser artiklen, hvor meget af heritabiliteten for en række træk kan forklares ved forskellige SNPer (og hvor meget denne øges i takt med at den mindre alleltærskel sænkes). Artiklen angiver også opdaterede estimater for heritabilitetsberigelser, idet den viser den stærke indvirkning, som bevarede områder og kodningsområder har på en bred vifte af komplekse træk og sygdomme.
Artiklen tilvejebringer ligeledes en detaljeret vejledning til forskellige metoder til SNP-baserede heritabilitetsanalyser. Den forklarer de mange måder, hvorpå disse analyser gør os i stand til bedre at forstå komplekse træk. Eksempelvis kan de bruges til at anslå den totale fordeling af SNPer, til at finde de vigtigste områder af genomet og til at måle indvirkningen af selektionstryk. Vejledningen beskriver både metoder, der anvender data på individniveau og de metoder, der bruger opsummerende statistikker.
I en kommentar til artiklen siger Doug Speed "I denne artikel har vi forsøgt at forklare, hvilke fremskridt der er sket på ti år inden for området for SNP-baseret heritabilitetsanalyse. De oprindelige metoder, der blev introduceret i 2010, var tilpasninger af analyser designet til dyre- og planteavl. Selvom disse metoder gav vigtige indblik i komplekse træk, var de grundlæggende set fejlbehæftede, fordi de var baserede på uhensigtsmæssige modelleringsantagelser. Vores nye studie viser først, hvordan nye metoder og modeller er blevet udviklet for at adressere disse fejl. Dernæst beskriver vi de nyeste resultater inden for komplekse træks genetiske arkitekturer, herunder hvordan disse træk er blevet formet ved at finpudse selektionen".
Supplerende oplysninger | |
Vi bestræber os på, at alle vores artikler lever op til Danske Universiteters principper for god forskningskommunikation. På den baggrund er artiklen suppleret med følgende oplysninger: | |
Studietype | Dataanalyser |
Finansiering
| This research has been conducted using the UK Biobank Resource under Application Number 45918. Doug Speed is supported by the European Union Horizon 2020 Research and Innovation Programme under the Marie Skłodowska-Curie grant 754513, by Aarhus University Research Foundation (AUFF), by the Independent Research Fund Denmark under Project no. 702500094B, and by a Lundbeck Foundation Experiment Grant. This research was partially supported by grant DP190103188 from the Australian Research Council to David Balding and Doug Speed. |
Samarbejdspartnere
| Anubhav Kaphle og David Balding School of Mathematics and Statistics, University of Melbourne, Australien |
Interessekonflikter
| Parterne erklærer, at der ikke er nogen interessekonflikt |
Læs mere
| SNP-based heritability and selection analyses: Improved models and new results Doug Speed, Anubhav Kaphle, David J. Balding. BioEssays. 2022. https://doi.org/10.1002/bies.202100170 |
Kontakt | Professor Doug Speed Mail.: doug@qgg.au.dk |
