Nye og forbedrede metoder til prædiktion af fænotyper skal bruges i personaliseret medicin
Professor Doug Speed fra Center for Kvantitativ Genetik og Genomforskning (QGG) og kolleger fra Bioinformatics Research Center (BiRC) og Center for Registerforskning (NCRR) ved Aarhus Universitet har udviklet forbedrede metoder til prædiktion af komplekse træk. Studiet blev offentliggjort i denne uge i tidsskriftet Nature Communications.
Der er stor interesse for muligheden af at bruge et menneskes genetiske information til at prædiktere dets fænotyper. Dette er særligt vigtigt indenfor personaliseret medicin, der har som mål at kunne prædiktere præcist, hvilke personer der vil udvikle specifikke sygdomme eller vil kunne have fordel af en specifik medicin.
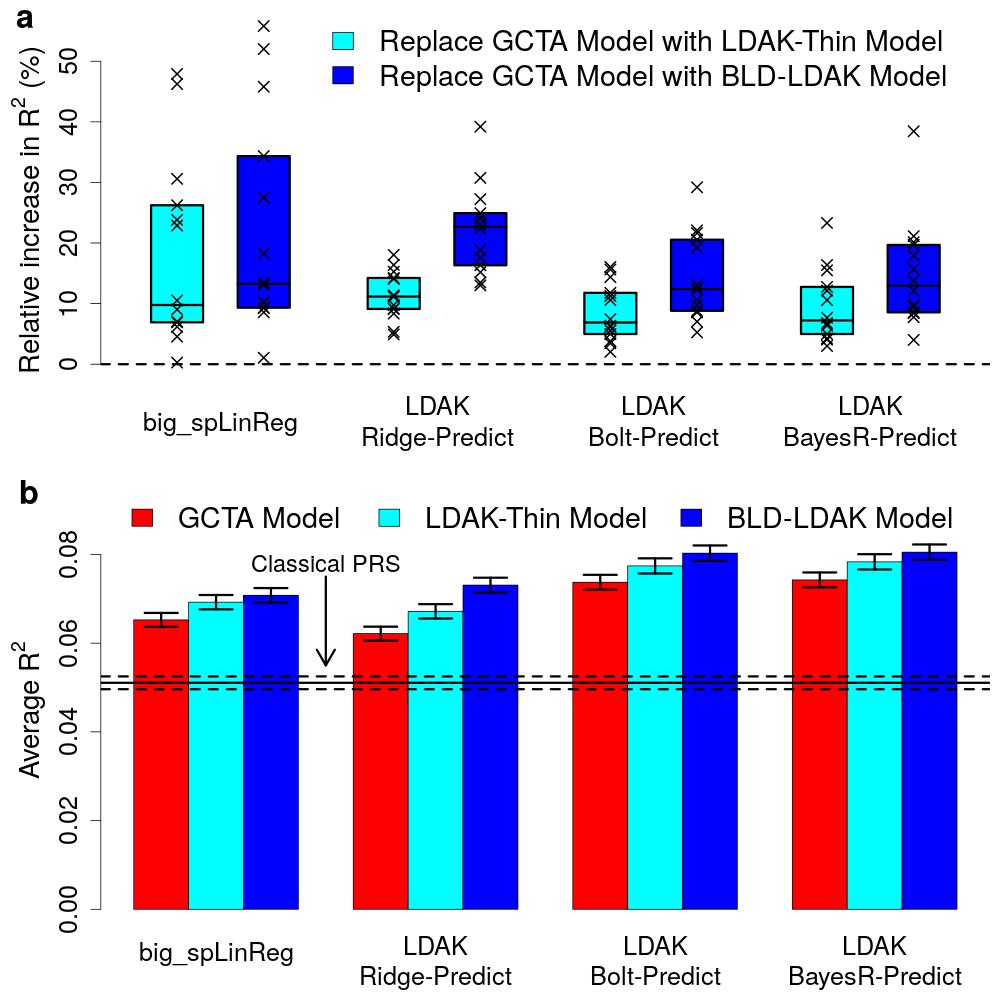
Doug Speed og hans kolleger observerede, at de fleste eksisterende prædiktionsværktøjer antager, at hver enkelt genetisk variant er lige vigtig. Denne antagelse er ikke optimal, idet nylige arbejder har vist, at en variant vigtighed afhænger af faktorer såsom dens frekvens, lokale niveauer af ubalance mellem bindinger (linkage disequilibrium) og funktionelle annotationer. Denne nye artikel præsenterer derfor otte nye prædiktionsværktøjer der tillader alternative antagelser og viser, at dette muliggør substantielt forbedret prædiktion på tværs af en bred vifte af træk.
Fire af de nye værktøjer anvender data på individniveau. Artiklen viser at det bedste af værktøjerne, LDAK-Bolt-Predict, udkonkurrerer de eksisterende værktøjer Lasso, BLUP, Bolt-LMM og BayesR for alle 14 undersøgte fænotyper. De resterende fire nye værktøjer anvender beskrivende statistikker (summary statistics). Artiklen viser, at det bedste af disse, LDAK-BayesR-SS, udkonkurrerer de eksisterende værktøjer lassosum, sBLUP, LDpred og SBayesR for 223 af de 225 fænotyper, der blev undersøgt. I gennemsnit udkonkurrerede de nye værktøjer de eksisterende med 14% (sd 1), svarende til at øge datamængden med ca 25%.
- ‘For at personaliseret medicin kan blive en realitet har vi brug for modeller, der præcist kan prædiktere en persons fænotyper baseret på personens genetiske information. Dette studie tilvejebringer statistiske værktøjer til udarbejdelse af genetiske prædiktionsmodeller, der er væsentligt mere præcise end eksisterende værktøjer’, forklarer Doug Speed og fortsætter:
- ‘Vores studie vil give en omgående fordel da det betyder, at vi nu bedre kan identificere personer, der har høj risiko for at udvikle forskellige sygdomme.’
Læs mere om de nye værktøjer i artiklen Improved genetic prediction of complex traits from individual-level data or summary statistics, og afprøv dem i softwarepakkerne LDAK og bigstatsr.
ITEM | INDHOLD OG FORMÅL |
Studietype | Først udviklede vi nye statistikværktøjer, dernæst sammenlignede vi deres ydeevne ved at anvende data fra UK Biobank, et populationsbaseret studie af cirka 500.000 individer registreret med en bred vifte af forskellige fænotyper. Vi beviste, at vores nye værktøjer udkonkurrerede eksisterende værktøjer for 223 ud af 225 undersøgte fænotyper. |
Eksterne samarbejdspartnere | Ingen |
Ekstern finansiering | Aarhus Universitets Forskningsfond, Lundbeckfonden, EU Horizon 2020 Research and Innovation Programme, Danmarks Frie Forskningsfond. |
Interessekonflikt | Forfatterne har erklæret ikke at have konkurrerende interesser. |
| Andet | Artiklen er fagfællebedømt. |
Link til videnskabelig artikel | Læs mere om de nye værktøjer her: Zhang Q, Privé F, Vilhjálmsson B, Speed D. Improved genetic prediction of complex traits from individual-level data or summary statistics. Nature Communications. |
Kontakt | Professor Doug Speed // doug@qgg.au.dk // Tlf.: +44 77538 26477 |
