Nyt værktøj til genbaseret associationsanalyse er mere præcist end lignende metoder
I en netop publiceret artikel har postdoc Takiy Berrandou og professor Doug Speed fra Center for Kvantitativ Genetik og Genomforskning (QGG), sammen med professor David Balding (University of Melbourne), foreslået LDAK-GBAT som et nyt værktøj til at udføre genbaseret associationsanalyse ved alene at anvende resultater fra et GWAS og et referencepanel af gendata.
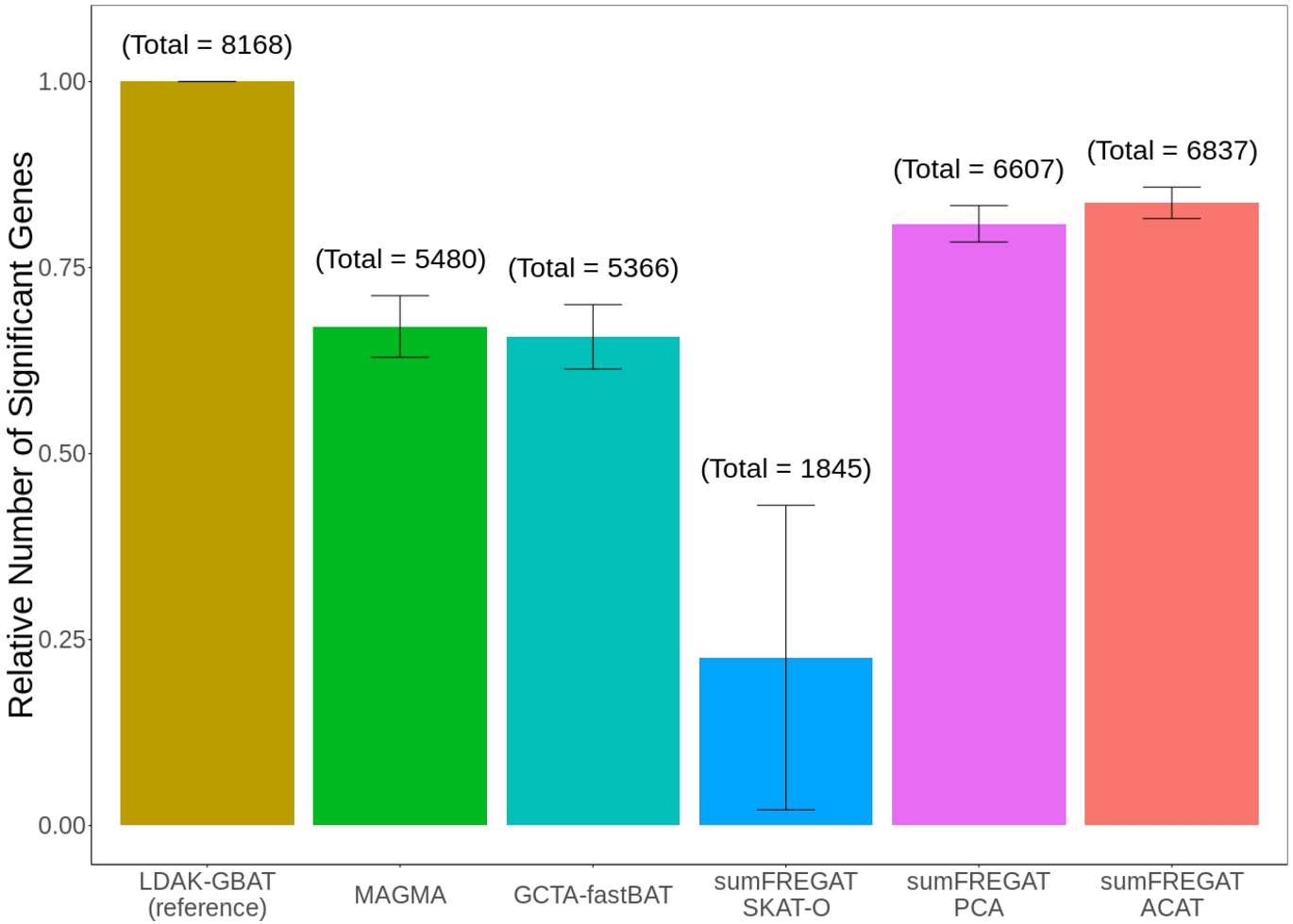
Deres artikel forklarer først setuppet og funktionerne ved LDAK-GBAT og bruger derefter simulerede data til at demonstrere nøjagtigheden og beregningseffektiviteten ved det nye værktøj. Dernæst sammenlignes LDAK-GBAT med fem andre genbaserede associationsanalysemetoder, herunder de mest anvendte metoder, MAGMA og GCTA-FastBAT, ved at anvende data fra 109 fænotyper fra UK Biobank, Million Veteran Program og Psychiatric Genomics Consortium. Forskerne fandt, at LDAK-GBAT er mere effektiv end de nuværende, tilgængelige værktøjer, og at det finder 20% flere signifikante, associerede gener end den næstbedste metode
Postdoc Takiy Berrandou, der er artiklens hovedforfatter, siger: "Udover at være beregningseffektiv og kraftfuld, indeholder LDAK-GBAT også et nyt clumping værktøj, som kan bruges til at prioritere associerede gener." Han fortsætter: "For eksempel fandt vores analyse 1511 gener, som er signifikant associerede med udvikling af Type 1 Diabetes. Derefter besluttede clumping værktøjet, hvilke af de 533 gener, som sandsynligvis er mest vigtige”.
Artiklen kan ses i det aktuelle nummer af American Journal of Human Genetics.
